그래디언트 부스팅 회귀 트리
- 여러 개의 결정 트리를 묶어 강력한 모델을 만드는 앙상블 기법 중 하나.
- 이름은 회귀지만 회귀와 분류에 모두 사용 가능
- 장점
- 지도학습에서 가장 강력함.
- 가장 널리 사용하는 모델 중의 하나
- 특성의 스케일 조정이 불필요 -> 정규화 불필요.
- 단점
- 매개변수를 잘 조정해야 한다는 것.
- 긴 훈련 시간.
- 트리 기반 모델을 사용. -> 희소한 고차원 데이터는 부적합. (특성이 많고 값이 별로 없는 데이터셋)
매개변수
- n_estmators: 트리의 개수 지정
- 너무 클 경우 모델이 복잡해지고 과대적합 가능성
- learning_rate: 이전 트리의 오차 보정 강도 조절.
- 메모리 한도에서 n_estimators 부터 설정.
- 이후 적절한 learning_rate 설정.
- 학습률이 크면 트리를 강하게 보정하여 복잡한 모델 생성. → 학습률을 오히려 낮추는 것이 좋음.
- max_depth: 각 트리의 복잡도를 낮추는 매개변수.
- 깊이는 5보다 깊어지지 않는 게 좋음.
GredientBoostingClassifier 사용하기
- 유방암 데이터셋 활용
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.ensemble import GradientBoostingClassifier
cancer = load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(
cancer.data, cancer.target, random_state = 0)
gbrt = GradientBoostingClassifier(random_state = 0)
gbrt.fit(X_train, y_train)
print("훈련 세트 정확도: {:.3f}".format(gbrt.score(X_train, y_train)))
print("테스트 세트 정확도: {:.3f}".format(gbrt.score(X_test, y_test)))
과대적합 상태.
gbrt = GradientBoostingClassifier(random_state = 0, max_depth= 1)
gbrt.fit(X_train, y_train)
print("훈련 세트 정확도: {:.3f}".format(gbrt.score(X_train, y_train)))
print("테스트 세트 정확도: {:.3f}".format(gbrt.score(X_test, y_test)))
gbrt = GradientBoostingClassifier(random_state = 0, learning_rate = 0.01)
gbrt.fit(X_train, y_train)
print("훈련 세트 정확도: {:.3f}".format(gbrt.score(X_train, y_train)))
print("테스트 세트 정확도: {:.3f}".format(gbrt.score(X_test, y_test)))
def plot_feature_importances_cancer(model):
n_features = cancer.data.shape[1]
plt.barh(np.arange(n_features), model.feature_importances_, align="center")
plt.yticks(np.arange(n_features), cancer.feature_names)
plt.xlabel("feature_important")
plt.ylabel("feature")
plt.ylim(-1, n_features)gbrt = GradientBoostingClassifier(random_state = 0, max_depth= 1)
gbrt.fit(X_train, y_train)
plot_feature_importances_cancer(gbrt)
학습 데이터셋
# from preamble import *
from sklearn.model_selection import train_test_split
from sklearn.datasets import make_moons
from sklearn.datasets import load_breast_cancer
Xm, ym = make_moons(n_samples = 100, noise = 0.25, random_state = 3)
Xm_train, Xm_test, ym_train, ym_test = train_test_split(
Xm, ym, stratify = ym, random_state = 42)
cancer = load_breast_cancer()
Xc_train, Xc_test, yc_train, yc_test = train_test_split(
cancer.data, cancer.target, random_state = 0)
배깅(Bagging)
- 중복을 허용한 랜덤 샘플링으로 만든 훈련 세트를 사용하여 분류기를 각기 다르게 학습.
from sklearn.linear_model import LogisticRegression
from sklearn.linear_model import LinearRegression
from sklearn.ensemble import BaggingClassifier
bagging = BaggingClassifier(LogisticRegression(solver = 'liblinear'),
n_estimators = 100, oob_score = True, n_jobs = -1, random_state = 42)
bagging.fit(Xc_train, yc_train)
print("훈련 세트 정확도: {:.3f}".format(bagging.score(Xc_train, yc_train)))
print("테스트 세트 정확도: {:.3f}".format(bagging.score(Xc_test, yc_test)))
print("OOB 셈플의 정확도: {:.3f}".format(bagging.oob_score_))
from sklearn.tree import DecisionTreeClassifier
bagging = BaggingClassifier(DecisionTreeClassifier(), n_estimators = 5,
n_jobs = -1, random_state = 42)
bagging.fit(Xm_train, ym_train)
fig, axes = plt.subplots(2, 3, figsize = (20, 10))
for i, (ax, tree) in enumerate(zip(axes.ravel(), bagging.estimators_)):
ax.set_title("Tree {}".format(i))
mglearn.plots.plot_tree_partition(Xm, ym, tree, ax = ax)
mglearn.plots.plot_2d_separator(bagging, Xm, fill = True, ax = axes[-1, -1], alpha = .4)
axes[-1, -1].set_title("Bagging")
mglearn.discrete_scatter(Xm[:, 0], Xm[:, 1], ym)
plt.show()
bagging = BaggingClassifier(DecisionTreeClassifier(), n_estimators = 100,
oob_score = True, n_jobs = -1, random_state = 42)
bagging.fit(Xc_train, yc_train)
print("훈련 세트 정확도: {:.3f}".format(bagging.score(Xc_train, yc_train)))
print("테스트 세트 정확도: {:.3f}".format(bagging.score(Xc_test, yc_test)))
print("OOB 셈플의 정확도: {:.3f}".format(bagging.oob_score_))
엑스트라 트리(Extra-Trees)
- 후보 특성을 무작위로 분할한 다음 최적의 분할을 찾음.
from sklearn.ensemble import ExtraTreesClassifier
xtree = ExtraTreesClassifier(n_estimators = 5, n_jobs = -1, random_state = 0)
xtree.fit(Xm_train, ym_train)
fig, axes = plt.subplots(2, 3, figsize = (20, 10))
for i, (ax, tree) in enumerate(zip(axes.ravel(), xtree.estimators_)):
ax.set_title("tree {}".format(i))
mglearn.plots.plot_tree_partition(Xm, ym, tree, ax = ax)
mglearn.plots.plot_2d_separator(xtree, Xm, fill = True, ax = axes[-1, -1], alpha = .4)
axes[-1, -1].set_title("Extra tree")
mglearn.discrete_scatter(Xm[:, 0], Xm[:, 1], ym)
plt.show()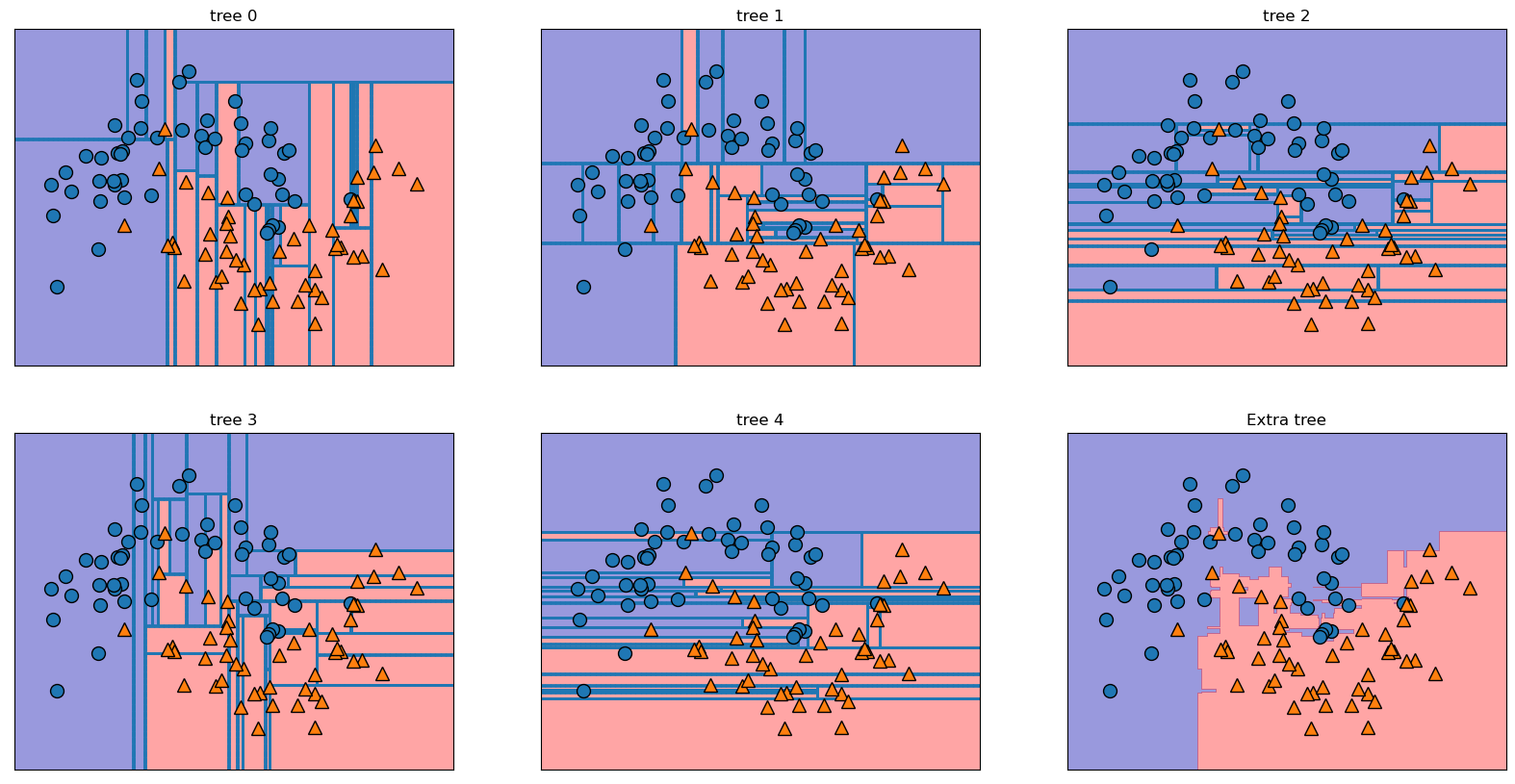
xtree = ExtraTreesClassifier(n_estimators = 100, n_jobs = -1, random_state = 0)
xtree.fit(Xc_train, yc_train)
print("훈련 세트 정확도: {:.3f}".format(xtree.score(Xc_train, yc_train)))
print("테스트 세트 정확도: {:.3f}".format(xtree.score(Xc_test, yc_test)))
n_features = cancer.data.shape[1]
plt.barh(range(n_features), xtree.feature_importances_, align="center")
plt.yticks(np.arange(n_features), cancer.feature_names)
plt.xlabel("Feature important")
plt.ylabel("Feature")
plt.ylim(-1, n_features)
plt.show()
에이다부스트(AdaBoost)
- 그레디언트 부스팅과는 달리 이전의 모델이 잘못 분류한 샘플에 가중치를 높여 다음 모델을 훈련.
from sklearn.ensemble import AdaBoostClassifier
ada = AdaBoostClassifier(n_estimators = 5, random_state = 42)
ada.fit(Xm_train, ym_train)
fig, axes = plt.subplots(2, 3, figsize = (20, 10))
for i, (ax, tree) in enumerate(zip(axes.ravel(), ada.estimators_)):
ax.set_title("Tree {}".format(i))
mglearn.plots.plot_tree_partition(Xm, ym, tree, ax = ax)
mglearn.plots.plot_2d_separator(ada, Xm, fill = True, ax = axes[-1, -1], alpha = .4)
axes[-1, -1].set_title("AdaBoost")
mglearn.discrete_scatter(Xm[:, 0], Xm[:, 1], ym)
plt.show()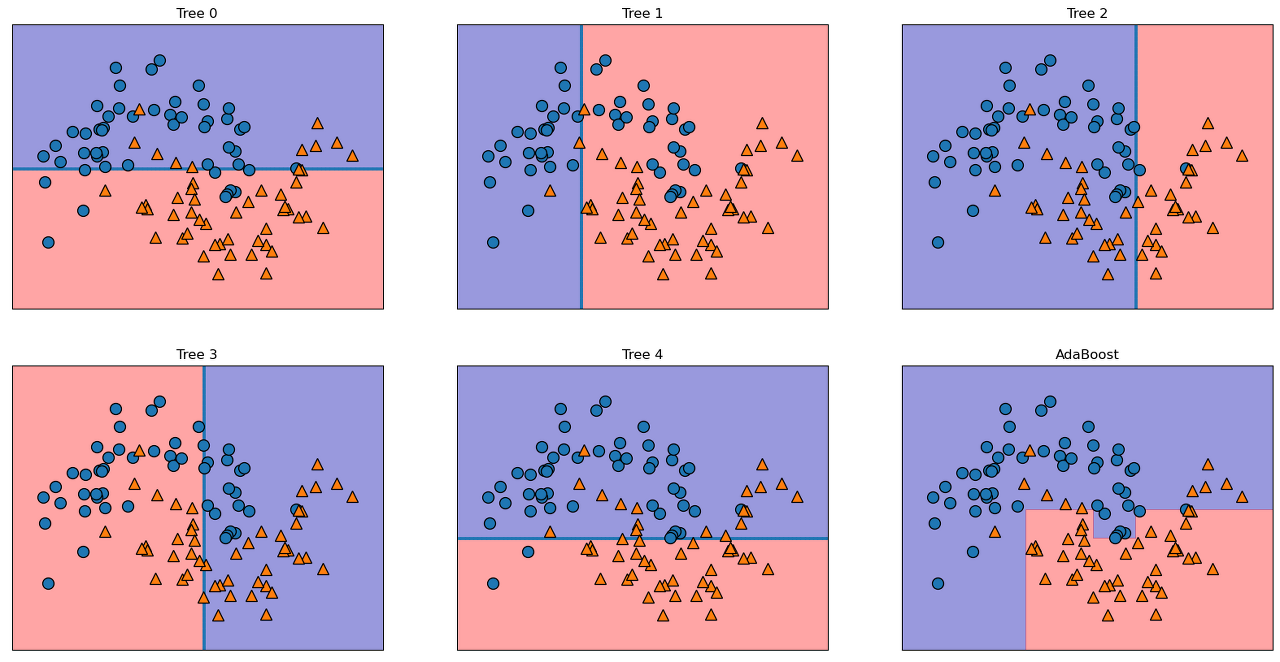
ada = AdaBoostClassifier(n_estimators = 100, random_state = 42)
ada.fit(Xc_train, yc_train)
print("훈련 세트 정확도: {:.3f}".format(ada.score(Xc_train, yc_train)))
print("테스트 세트 정확도: {:.3f}".format(ada.score(Xc_test, yc_test)))
plt.barh(range(n_features), ada.feature_importances_, align = "center")
plt.yticks(np.arange(n_features), cancer.feature_names)
plt.xlabel("Feature important")
plt.ylabel("Feature")
plt.ylim(-1, n_features)
plt.show()
히스토그램 기반 부스팅
from sklearn.ensemble import HistGradientBoostingClassifier
hgb = HistGradientBoostingClassifier(random_state = 42)
hgb.fit(Xm_train, ym_train)
mglearn.plots.plot_2d_separator(hgb, Xm, fill = True, alpha = .4)
plt.title("HistGradientBoosting")
mglearn.discrete_scatter(Xm[:, 0], Xm[:, 1], ym)
plt.show()

hgb = HistGradientBoostingClassifier(random_state = 42)
hgb.fit(Xc_train, yc_train)
print("훈련 세트 정확도: {:.3f}".format(hgb.score(Xc_train, yc_train)))
print("테스트 세트 정확도: {:.3f}".format(hgb.score(Xc_test, yc_test)))
from sklearn.inspection import permutation_importance
result = permutation_importance(hgb, Xc_train, yc_train,
n_repeats = 10, random_state = 42, n_jobs = -1)
plt.barh(range(n_features), result.importances_mean, align="center")
plt.yticks(np.arange(n_features), cancer.feature_names)
plt.xlabel("Feature important")
plt.ylabel("Feature")
plt.ylim(-1, n_features)
plt.show()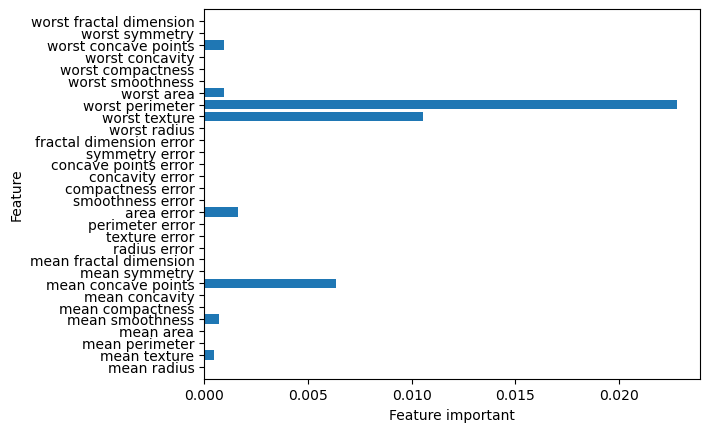
커널 서포트 백터 머신 (SVM)
- 입력 데이터에서 단순한 초평면으로 정의되지 않는 더 복잡한 모델을 만들 수 있도록 확장한 지도 학습 모델.
from sklearn.datasets import make_blobs
X, y = make_blobs(centers = 4, random_state = 8)
y = y % 2
mglearn.discrete_scatter(X[:, 0], X[:, 1], y)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
분류를 위한 선형 모델은 직선으로만 데이터 포인트를 나눌 수 있어서 이런 데이터셋에서는 맞지 않음.
from sklearn.svm import LinearSVC
linear_svm = LinearSVC(max_iter = 5000, tol=1e-3).fit(X, y)
mglearn.plots.plot_2d_separator(linear_svm, X)
mglearn.discrete_scatter(X[:, 0], X[:, 1], y)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
X_new = np.hstack([X, X[:, 1:] ** 2])
from mpl_toolkits.mplot3d import Axes3D, axes3d
figure = plt.figure()
# 3차원 그래프
if matplotlib.__version__ >= "3.4":
# Axes3D가 자동으로 그램에 추가되는 방식은 matplotlib 3.4 버전에서
# deprecated 되었습니다.
# 이와 관련된 경고를 피하려면 auto_add_to_figure = False로 지정하고
# figure.add_axes(ax)로 직접 추가하세요.
ax = Axes3D(figure, elev = - 512, azim = -26, auto_add_to_figure = False)
figure.add_axes(ax)
else:
ax = Axes3D(figure, elev = -152, azim = -26)
mask = y == 0
ax.scatter(X_new[mask, 0], X_new[mask, 1], X_new[mask, 2], c='b',
cmap = mglearn.cm2, s = 60, edgecolor = 'k')
ax.scatter(X_new[~mask, 0], X_new[~mask, 1], X_new[~mask, 2], c='b', marker = '^',
cmap = mglearn.cm2, s = 60, edgecolor = 'k')
ax.set_xlabel("Feature 0")
ax.set_ylabel("Feature 1")
ax.set_zlabel("Feature 1 ** 2")
linear_svm_3d = LinearSVC(max_iter = 5000).fit(X_new, y)
coef, intercept = linear_svm_3d.coef_.ravel(), linear_svm_3d.intercept_
# 선형 결정 경계 그리기
figure = plt.figure()
if matplotlib.__version__ >= '3.4':
# Axes3D가 자동으로 그램에 추가되는 방식은 matplotlib 3.4 버전에서
# deprecated 되었습니다.
# 이와 관련된 경고를 피하려면 auto_add_to_figure = False로 지정하고
# figure.add_axes(ax)로 직접 추가하세요.
ax = Axes3D(figure, elev = - 512, azim = -26, auto_add_to_figure = False)
figure.add_axes(ax)
else:
ax = Axes3D(figure, elev = -152, azim = -26)
xx = np.linspace(X_new[:, 0].min() - 2, X_new[:, 0].max() + 2, 50)
yy = np.linspace(X_new[:, 1].min() - 2, X_new[:, 1].max() + 2, 50)
XX, YY = np.meshgrid(xx, yy)
ZZ = (coef[0] * XX + coef[1] * YY + intercept) / -coef[2]
ax.plot_surface(XX, YY, ZZ, rstride = 8, cstride = 8, alpha = 0.3)
ax.scatter(X_new[mask, 0], X_new[mask, 1], X_new[mask, 2], c='b',
cmap = mglearn.cm2, s = 60, edgecolor = 'k')
ax.scatter(X_new[~mask, 0], X_new[~mask, 1], X_new[~mask, 2], c='r', marker = '^',
cmap = mglearn.cm2, s = 60, edgecolor = 'k')
ax.set_xlabel("Feature 0")
ax.set_ylabel("Feature 1")
ax.set_zlabel("Feature 1 ** 2")
ZZ = YY ** 2
dec = linear_svm_3d.decision_function(np.c_[XX.ravel(), YY.ravel(), ZZ.ravel()])
plt.contourf(XX, YY, dec.reshape(XX.shape), levels = [dec.min(), 0, dec.max()],
camap = mglearn.cm2, alpha = 0.5)
mglearn.discrete_scatter(X[:, 0], X[:, 1], y)
ax.set_xlabel("Feature 0")
ax.set_ylabel("Feature 1")
커널 기법
- 데이터셋에 비선형 특성을 추가하여 강력한 선형 모델 생성 가능
- 어떤 특성을 추가해야할지 파악하기 어려운 문제
- 특성을 지나치게 추가할 경우 연산 비용 증가 문제.
- 실제 데이터를 확장하지 않고 확장된 특성에 대한 데이터 포인트들의 거리 계산.
from sklearn.svm import SVC
X, y = mglearn.tools.make_handcrafted_dataset()
svm = SVC(kernel = 'rbf', C = 10, gamma = 0.1).fit(X, y)
mglearn.plots.plot_2d_separator(svm, X, eps=.5)
# 데이터 포인터 그리기
mglearn.discrete_scatter(X[:, 0], X[:, 1], y)
# 서포트 백터
sv = svm.support_vectors_
# dual_coef_의 부호에 의해 서포트 백터의 클래스 레이블이 결정됩니다.
sv_labels = svm.dual_coef_.ravel() > 0
mglearn.discrete_scatter(sv[:, 0], sv[:, 1], sv_labels, s = 15, markeredgewidth = 3)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
fig, axes = plt.subplots(3, 3, figsize = (15, 10))
for ax, C in zip(axes, [-1, 0, 3]):
for a, gamma in zip (ax, range(-1, 2)):
mglearn.plots.plot_svm(log_C = C, log_gamma = gamma, ax = a)
axes[0, 0].legend(['클래스 0', '클래스 1', '클래스 0 서포트 백터',
'클래스 1 서포트 백터'], ncol = 4, loc = (.9, 1.2))
X_train, X_test, y_train, y_test = train_test_split(
cancer.data, cancer.target, random_state = 0)
svc = SVC()
svc.fit(X_train, y_train)
print("훈련 세트 정확도: {:.3f}".format(svc.score(Xc_train, yc_train)))
print("테스트 세트 정확도: {:.3f}".format(svc.score(Xc_test, yc_test)))
plt.boxplot(X_train, manage_ticks = False)
plt.yscale('symlog')
plt.xlabel("Feature list")
plt.ylabel("Feature width")
# 훈련 세트에서 특성별 최솟값 계산
min_on_training = X_train.min(axis = 0)
# 훈련 세트에서 특성별 (최댓값 - 최솟값) 범위 계산
range_on_training = (X_train - min_on_training).max(axis = 0)
# 훈련 데이터에 최솟값을 빼고 범위로 나누면
# 각 특성에 대해 최솟값은 0 최댓값은 1이다.
X_train_scaled = (X_train - min_on_training) / range_on_training
print("특성별 최솟값\n", X_train_scaled.min(axis = 0))
print("특성별 최댓값\n", X_train_scaled.max(axis = 0))
# 테스트 세트에도 같은 작업을 적용하지만
# 훈련 세트에서 계산한 최솟값고 범위를 사용합니다.
X_test_scaled = (X_test - min_on_training) / range_on_training
svc = SVC()
svc.fit(X_train_scaled, y_train)
print("훈련 세트 정확도: {:.3f}".format(svc.score(X_train_scaled, y_train)))
print("테스트 세트 정확도: {:.3f}".format(svc.score(X_test_scaled, y_test)))
svc = SVC(C = 20)
svc.fit(X_train_scaled, y_train)
print("훈련 세트 정확도: {:.3f}".format(svc.score(X_train_scaled, y_train)))
print("테스트 세트 정확도: {:.3f}".format(svc.score(X_test_scaled, y_test)))
'ABC 부트캠프 > 머신러닝' 카테고리의 다른 글
| 19일차 - 지도학습 2 (1) | 2023.04.12 |
|---|---|
| 18일차 - 지도학습 1 (0) | 2023.04.11 |
| 17일차 - 머신러닝(ML) (0) | 2023.04.11 |



댓글